Kontakt: magda.fikus@gmail.com
Idea przyglądania się strukturze przestrzennej biocząsteczek pojawiła się dość dawno, na przykład w rozważaniach o enzymach i substratach (model zamka i klucza). Sama po raz pierwszy uruchomiłam biochemiczną wyobraźnię przestrzenną, patrząc na model DNA zrobiony z drucików przedstawiony przez Jamesa Watsona i Francisa Cricka w 1953 roku. Uprzednio wystarczało mi przyswajanie z podręczników biochemii dwuwymiarowych obrazów cząsteczek wchodzących ze sobą w oddziaływania.
Badania procesów zwijania się biopolimerów w określone struktury przestrzenne musiały być poprzedzone uzyskaniem wiedzy o ich budowie chemicznej. Z jakich podjednostek składają się DNA, RNA i białka i jak są one połączone w nić? W polimerze kolejne podjednostki łączą się ze sobą chemicznymi wiązaniami. Jednocześnie długa nić może się zwijać w bardzo zwarte struktury dzięki „bocznym” oddziaływaniom elektrostatycznym między różnymi podjednostkami. Często taka struktura przestrzenna ze względu na energię oddziaływania jest dla danego polimeru optymalna, a więc powtarzalna. Opracowano wiele metod na oznaczenie kształtów przestrzennych biopolimerów, a uzyskanie ich detali stało się możliwe, czasami nawet rutynowe, w naszym XXI wieku.
Problem kształtu zwiniętych struktur nici polimerów był szczegółowo badany w przypadku białek – polimerów składających się z aminokwasów. Kilkanaście lat temu dzięki poznaniu budowy chemicznej wielu białek i wiedzy o ich zwijaniu się w określone, powtarzalne struktury podjęto szerokie badania w celu uzyskania możliwości przewidywania tych struktur. Postęp nastąpił po wprowadzeniu do tej dziedziny metod bioinformatycznych. Duże zasługi położyło tu kilka polskich zespołów biologów molekularnych. Okazało się, że tylko zwinięte w określony, przewidywalny sposób białka wykazują aktywność biologiczną.
W przypadku RNA zauważono współcześnie podobne zjawiska w grupie koronawirusów. Wśród nich wyróżniają się podgatunki typu beta, ze względu na patogenność oraz wywołanie epidemii oraz globalnej pandemii. Rozpoznanie wspólnych cech struktury tej grupy wirusów może przyczynić się do szybszej diagnozy i skutecznej terapii wirusów już poznanych i tych, które pojawią się w przyszłości.
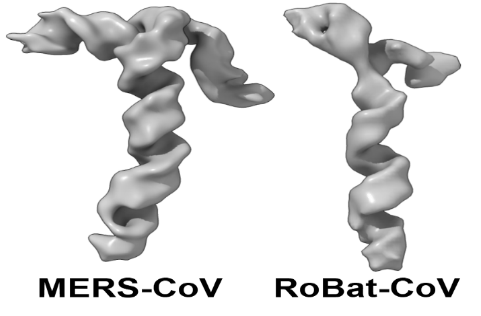
Genomami koronawirusów są jednoniciowe cząsteczki RNA o długości około 30 tys. podjednostek, nukleotydów. Kolejność (sekwencja) ułożenia nukleotydów (4 rodzaje) stanowi informację genetyczną wirusa. Początkowych 500 nukleotydów, o różnorodnej sekwencji w różnych wirusach, wykazuje jednak (czego nie oczekiwano) pewne cechy wspólne. Region 500 nukleotydów pełni podstawową rolę w procesie namnażania wirusa w zakażonej komórce, w obszarze tej sekwencji rozpoczyna się synteza kodowanych przez wirusa białek. W międzynarodowym zespole naukowców z Warszawy, Krakowa i Madrytu (liderem projektu jest prof. Janusz Bujnicki z Międzynarodowego Instytutu Biologii Komórkowej i Molekularnej (IIMCB) w Warszawie) odkryto, że ten odcinek RNA zwija się w różnych wirusach w podobne struktury modelowane z atomową rozdzielczością. Rozróżniane są odcinki zwinięte helikalnie i między nimi jednoniciowe odcinki łącznikowe. Wspomniane fragmenty RNA w czterech wirusach zanalizowano, stosując bogaty zestaw metod biochemicznych, biofizycznych, bioinformatycznych; w części badań wykonano pomiary zależności badanych struktur od temperatury – w podwyższonej rozpadają się helisy. Raz jeszcze zadziwiająca wydała się cecha znacznego podobieństwa wykrywanych struktur wobec znaczących rozbieżności w sekwencjach nukleotydów. Poza sekwencją nukleotydów, o aktywności układów biologicznych, decydującą rolę może odgrywać „kod” wyższego rzędu – struktura przestrzenna. Rozpoznanie takich struktur w koronawirusach może doprowadzić do nowych rozwiązań diagnostycznych i terapeutycznych. Oczywiście chcielibyśmy wiedzieć już teraz, co właściwie dzieje się w zakażonej komórce, co robią te strukturalne „haczyki” – poszukiwania trwają i w Polsce, i w Hiszpanii.
Odcinek RNA w różnych wirusach zwija się w podobne struktury. Fragmenty RNA w czterech wirusach poddanych analizie w badaniu międzynarodowego zespołu naukowców z Warszawy, Krakowa i Madrytu. Źródło: Międzynarodowy Instytut Biologii Molekularnej i Komórkowej w Warszawie